Éclairage sur l’origine des métastases
Une nouvelle approche prenant en compte la grande hétérogénéité et le phénotype des cellules tumorales permet de mieux cibler la recherche sur les métastases. Un gène prévenant leur apparition a été identifié par une équipe de l’UNIGE.
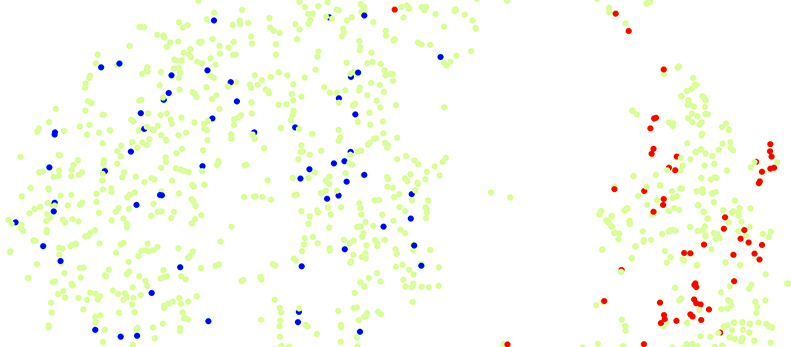
Le séquençage d’ARN avec la technique du Spiked-single-cell attribue des phénotypes à des groupes de cellules. Exemple de cartographie d’un seul groupe de cellules montrant (en vert) la population hétérogène de cellules cancéreuses qui se divise en deux groupes. La cartographie des pointages permet d’identifier les cellules ayant un potentiel métastatique variable par le retour du clone métastatique (en rouge) vers un groupe, et du clone non métastatique (en bleu) vers l’autre groupe. © UNIGE/Ariel Ruiz i Altaba
Pour développer des traitements efficaces, il faut être capable de comprendre l’effet spécifique d’une substance anticancéreuse sur le type de cellule, voire la cellule, donnant naissance aux métastases dans l’immense hétérogénéité cellulaire des tumeurs. Une équipe de l’Université de Genève (UNIGE) a utilisé une technique d’avant-garde, appelée spiked-scRNAseq, liant la transcriptomique aux phénotypes cellulaires des tumeurs du cancer du côlon. L’importance du gène VSIG1, impliqué dans les interactions intercellulaires, a pu être identifiée comme un facteur préventif des métastases. Au-delà de cette découverte porteuse d’espoir pour l’élaboration de futurs traitements, cette publication parue dans Cell Reports valide l’utilisation de cette technologie pour tester des médicaments actifs contre les métastases, y compris pour des approches personnalisées.
La plupart des traitements médicamenteux contre le cancer ne fonctionnent pas de manière optimale contre les métastases. Ils ont en effet été développés pour avoir une action globale et le plus grand effet possible sur la moyenne des patients. Or, pour être efficaces, «les drogues devraient être ciblées uniquement sur les cellules générant des métastases», indique le Professeur Ariel Ruiz i Altaba, chercheur à la faculté de Médecine de l’UNIGE. En effet, les tumeurs sont composées de cellules très hétérogènes: certaines vont donner lieu à des métastases alors que d’autres pas. Plus intéressant pour le développement de nouveaux traitements, des cellules tumorales vont freiner ou accélérer la croissance des métastases. Scientifiques et médecins cherchent depuis longtemps à savoir comment cibler le bon type de cellules tumorales pour vaincre le cancer.
L’équipe du Professeur Ruiz i Altaba a mis en place une approche permettant de définir les phénotypes des tumeurs, de les cloner et de les tracer grâce à une analyse à l’échelle cellulaire, tant du génome que de l’expression des ARN messagers qui en découlent. Pour rappel, les gênes d’une cellule, ou son génotype sont d’abord copiés en ARN messager qui est souvent utilisé pour la synthèse des protéines. Ces dernières sont donc l’expression visible des gènes, leurs actions définissent les caractéristiques mesurables de la cellule, soit son phénotype. «Notre approche permet d’identifier les pièces manquantes du puzzle d’une tumeur, en liant l’expression du génotype au phénotype cellulaire. En substance, nous voulons savoir comment les cellules deviennent métastatiques et d’où elles proviennent», indique le chercheur.
La validation d’un modèle
En développant et utilisant la technique dite du spiked-scRNAseq, qui combine transcriptomique et détermination du phénotype d’une seule cellule en la «pointant» dans une population, les chercheurs et chercheuses ont réussi à définir précisément la composition de la population cellulaire tumorale tout en déterminant leur phénotype à l’échelle cellulaire. Pour ce faire ils/elles ont d’abord étiqueté génétiquement plusieurs types de cellules tumorales afin de pouvoir observer leur devenir et plus particulièrement leur comportement métastatique. Une fois le phénotype connu, les cellules du même clone ont été pointées dans la population hétérogène de cellules primaires du cancer du côlon et soumises à un séquençage unicellulaire. Le profil moléculaire des cellules ayant un potentiel métastatique a ainsi été identifié par le repérage des cellules métastatiques pointées dans ce groupe de cellules.
Fort de cette approche, des drogues et leur effet antimétastasique ont pu être testés précisément. «En d’autres termes, nous pouvons utiliser cette approche pour analyser des composés pour leur action sur les cellules générant des métastases chez des patients individuels.», indique Marianna Silvano, postdoctorante à l’UNIGE et copremière auteure de l’étude.
Identification d’un gène répresseur de métastase
Les chercheurs ont ensuite appliqué cette approche pour comprendre le comportement tumoral. «Nous avons cherché à définir le statut pré métastatique d’une tumeur et d’évaluer son potentiel métastatique», ajoute-t-elle. Pour ce faire, l’équipe de recherche genevoise a découvert qu’en mélangeant des cellules ayant un phénotype métastatique avec des cellules non métastatiques, les premières stoppaient la migration de ces dernières. «C’est l’indication que des interactions cellulaires restreintes sont importantes dans le processus de formation des métastases», explique Ariel Ruiz i Altaba.
Forts de cet enseignement, analyses génomiques et transcriptomiques en main, l’équipe de recherche s’est focalisée sur les gènes impliqués dans les voies de signalisation importantes pour les interactions cellulaires. Carolina Bernal, postdoctorante et co-première auteure de l’étude, a identifié le gène VSIG1. Marianna Silvano et elle ont conclu qu’il était essentiel pour l’interaction restrictive entre les cellules tumorales non métastatiques et métastatiques. «En l’exprimant dans une cellule tumorale, on fait décroitre les métastases in vivo et in vitro. Si on l’enlève, on observe un accroissement des métastases», conclut Marianna Silvano. Les cellules métastatiques des tumeurs primaires peuvent être localisées grâce au spiked-scRNAseq, rendant possible l’identification de gènes essentiels. Des médicaments actifs pourront ainsi être identifiés pour développer de nouveaux traitements efficaces contre le cancer du côlon, comme pour d’autres cancers.
10 nov. 2020