Andrey lab: contrôle épigénétique du développement embryonnaire
Pendant l'embryogenèse, l'activation et la répression des gènes dans le temps et dans l'espace permettent la formation normale des tissus et des organes. Les processus moléculaires qui orchestrent ces activités sont donc essentiels au développement de l'organisme et leurs perturbations peuvent entraîner des malformations ou des maladies.
Konrad Guenther, Vom Urtier zum Menschen, 1909.
Dans notre laboratoire, nous étudions les processus moléculaires qui contrôlent l’expression des gènes lors du développement embryonnaire. Notre recherche se focalise sur deux axes principaux.
- Nous étudions comment les régions régulatrices communiquent avec leurs gènes cibles lors du développement embryonnaire. En particulier, nous voulons déterminer comment la proximité spatiale entre ces éléments est établie ainsi que la façon dont elle détermine l’activation transcriptonnelle des gènes et donc la formation des organes.
- Nous caractérisons l’identité transcriptonnelle ainsi que la structure génomique de populations cellulaires spécifiques lors du développement normal et anormal des organes.

Le Capture-HiC à haute résolution permet de définir l'organisation tridimensionelle du génome
Afin d’effectuer notre recherche, notre laboratoire, en collaboration avec la plateforme de transgénèse de la faculté de médecine, utilise la complémentation tétraploïde. Cette méthode permet la production d’embryons à partir de cellules souches murines de type sauvage ou génétiquement modifiées, tout en réduisant drastiquement le nombre d’animaux utilisés. Nous utilisons ensuite des méthodes de pointe pour déterminer, lors de l’organogénèse de ces embryons, les changements dans la structure de la chromatine et dans l’activité des gènes. Notre objectif final est d’établir les règles qui gouvernent les relations entre la chromatine, l’expression des gènes et le développement normal/anormal des embryons.
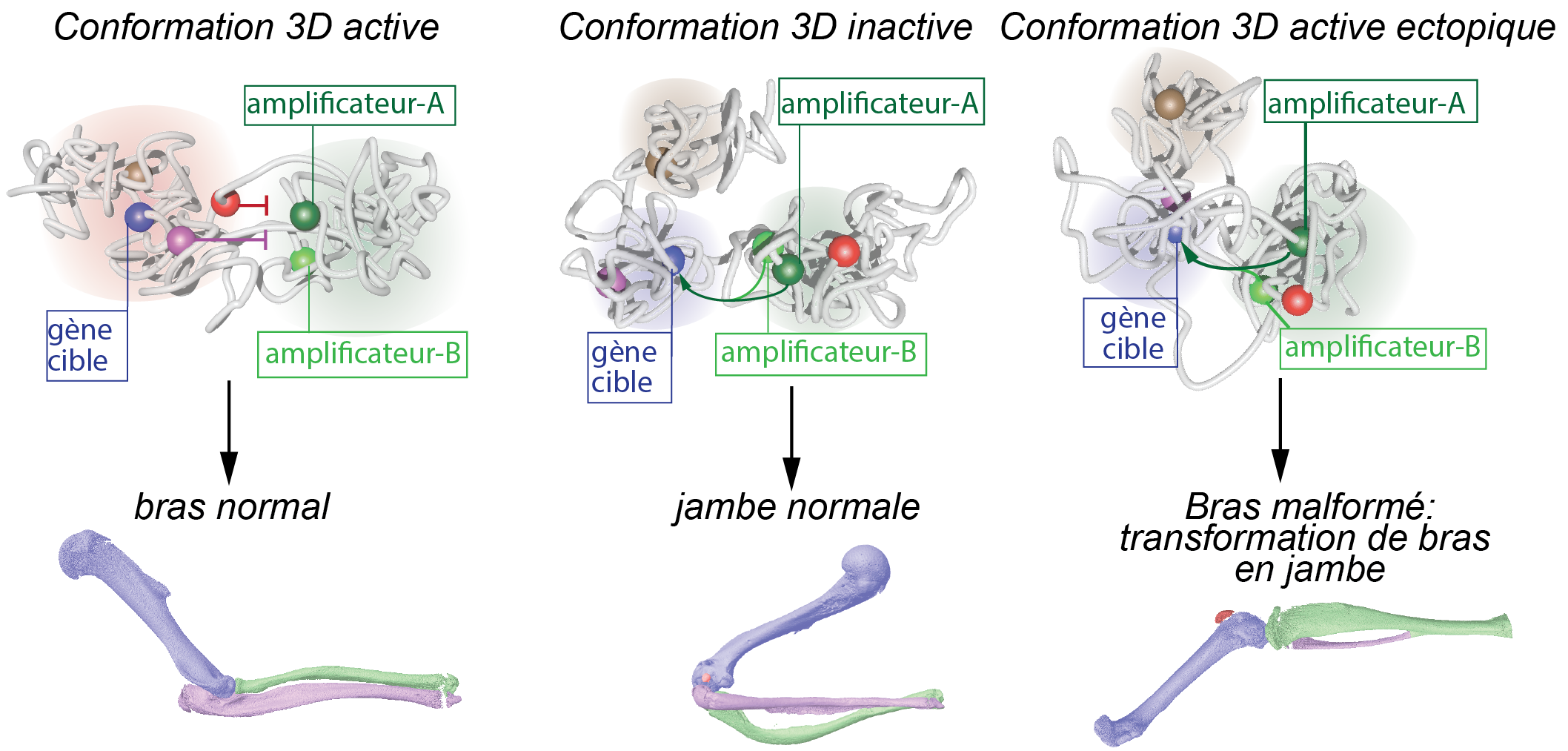
Certaines altérations dans la conformation 3D du génome mènent à l'expression ectopique de gènes et à des malformations des bras (adapté de Kragesteen et al., Nat Genet., 2018)
Publications Sélectionnées
Chondrogenic Enhancer Landscape of Limb and Axial Skeleton Development
Darbellay F., Ramisch A., Lopez-Delisle L., Koiscki M., Visel A. and Andrey G.
Preformed Chromatin Topology Assists Transcriptional Robustness of Shh during Limb Development.
Proc Natl Acad Sci U S A. 2019 May 30. pii: 201900672. doi: 10.1073/pnas.1900672116
Dynamic 3D chromatin architecture contributes to enhancer specificity and limb morphogenesis.
Kragesteen BK, Spielmann M, Paliou C, Heinrich V, Schöpflin R, Esposito A, Annunziatella C, Bianco S, Chiariello AM, Jerković I, Harabula I, Guckelberger P, Pechstein M, Wittler L, Chan WL, Franke M, Lupiáñez DG, Kraft K, Timmermann B, Vingron M, Visel A, Nicodemi M, Mundlos S, Andrey G.
Nat Genet. 2018 Oct;50(10):1463-1473. doi: 10.1038/s41588-018-0221-x. Epub 2018 Sep 27.
Characterization of hundreds of regulatory landscapes in developing limbs reveals two regimes of chromatin folding.
Andrey G, Schöpflin R, Jerković I, Heinrich V, Ibrahim DM, Paliou C, Hochradel M, Timmermann B, Haas S, Vingron M, Mundlos S.
Genome Res. 2017 Feb;27(2):223-233. doi: 10.1101/gr.213066.116. Epub 2016 Dec 6.
Formation of new chromatin domains determines pathogenicity of genomic duplications.
Franke M, Ibrahim DM, Andrey G, Schwarzer W, Heinrich V, Schöpflin R, Kraft K, Kempfer R, Jerković I, Chan WL, Spielmann M, Timmermann B, Wittler L, Kurth I, Cambiaso P, Zuffardi O, Houge G, Lambie L, Brancati F, Pombo A, Vingron M, Spitz F, Mundlos S.
Nature. 2016 Oct 13;538(7624):265-269. doi: 10.1038/nature19800. Epub 2016 Oct 5.
Deletions, Inversions, Duplications: Engineering of Structural Variants using CRISPR/Cas in Mice.
Kraft K, Geuer S, Will AJ, Chan WL, Paliou C, Borschiwer M, Harabula I, Wittler L, Franke M, Ibrahim DM, Kragesteen BK, Spielmann M, Mundlos S, Lupiáñez DG, Andrey G.
Cell Rep. 2015 Feb 4. pii: S2211-1247(15)00029-7. doi: 10.1016/j.celrep.2015.01.016.
A switch between topological domains underlies HoxD genes collinearity in mouse limbs.
Andrey G, Montavon T, Mascrez B, Gonzalez F, Noordermeer D, Leleu M, Trono D, Spitz F, Duboule D.
Science. 2013 Jun 7;340(6137):1234167. doi: 10.1126/science.1234167.
