Dossier/PRN
Voir les cellules comme on ne les a jamais vues
Le Pôle de recherche national «Biologie chimique» s’est fixé comme but d’étudier les processus biologiques qui se déroulent dans les cellules tout en gardant ces dernières vivantes. Pour parvenir à leurs fins, les chercheurs comptent développer des technologies innovantes
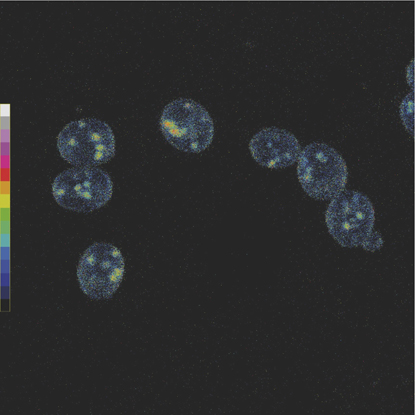
Ce qui se passe dans la cellule, brique essentielle des créatures vivantes, échappe encore largement aux sondes des biologistes. Les scientifiques savent qu’un nombre gigantesque de réactions biochimiques y ont leur siège et connaissent en général les différentes étapes qui les composent. Mais, dans leur grande majorité, elles demeurent invisibles et hors de contrôle. «Nous savons par exemple que, dans une cellule, le glucose est converti en pyruvate au cours d’une réaction appelée la glycolyse, explique Howard Riezman, professeur au Département de biochimie. Mais nous ignorons son organisation précise et nous ne pouvons en contrôler spécifiquement et efficacement aucune des étapes dans une cellule vivante. L’objectif du Pôle de recherche national (PRN) «Biologie chimique» que je dirige consiste à changer cet état des choses. Nous voulons rendre visibles et contrôlables les processus biologiques.»
Technologies innovantes
La stratégie des chercheurs du PRN est de tabler sur des technologies innovantes déjà existantes ainsi que sur celles qu’ils développeront eux-mêmes. Le pari est que les avancées techniques ont souvent permis à la science de réaliser de grands bonds en avant. «C’est cette orientation clairement affichée qui nous a probablement valu de figurer parmi les huit pôles choisis par la Confédération, estime Howard Riezman. Cette orientation devrait d’ailleurs favoriser l’éclosion au cours de prochaines années de plusieurs spin-off. Ces dernières pourront valoriser les innovations et les brevets qui ne manqueront pas d’être déposés en cours de route.»
Parmi les exemples de technologie ayant dopé la science figure la PCR, acronyme anglais pour réaction en chaîne par polymérase. Inventée dans les années 1980, cette technique permet de réaliser un nombre considérable de copies à partir d’un ou de quelques fragments d’ADN. Sa généralisation dans les laboratoires a bouleversé la recherche en génétique et ouvert la porte à d’innombrables découvertes.
Localiser des molécules
Autre exemple, plus récentw et plus proche des préoccupations actuelles de Howard Riezman: la découverte de la protéine fluorescente verte GFP. Empruntée à la méduse Aequorea victoria, cette molécule émet une lumière verte (après avoir été soumise à une lampe ultraviolette ou bleue). Depuis les années 1990, les scientifiques ont réussi à fusionner le gène de la GFP (et de ses multiples variantes développées depuis) avec les gènes d’autres protéines qu’ils désirent étudier. Alliée à des techniques de microscopie à haute résolution, cette manipulation leur permet de localiser dans la cellule toujours vivante la présence des molécules ainsi marquées et de mesurer à quel moment elles sont synthétisées.
Cependant Howard Riezman veut aller plus loin et pouvoir distinguer parmi les protéines qu’il veut étudier celles qui sont «actives» de celles qui sont «inactives». «Parmi les molécules que nous étudions, les kinases sont des enzymes qui jouent un rôle très important dans les cascades de réactions aussi appelées voies de signalisation, explique-t-il. On sait qu’à tout moment, dans une cellule, entre 90 et 95% de ces enzymes sont inactives, attendant qu’un signal vienne les réveiller. Un des projets que nous menons actuellement vise à développer une méthode permettant de visualiser uniquement les kinases actives, tout en laissant les autres dans l’ombre.»
Plus fort encore est l’opération consistant à activer, à volonté, une enzyme à un endroit précis de la cellule. Il peut s’agir d’une kinase – encore une – dont on veut étudier le comportement lorsqu’elle est accrochée à un microtubule, une fibre qui se forme en abondance notamment lors des phases de division cellulaire. «C’est possible, confirme Howard Riezman. Cela aussi fait d’ailleurs partie des projets du PRN.»
Pour parvenir à ses fins, un des outils les plus puissants dont s’apprête à se doter le PRN est la plateforme technologique ACCESS (pour Academic Chemical Screening Platform for Switzerland). Cette dernière ressemblera avant tout à une collection inédite de molécules de toutes formes et de toutes sortes, qu’elles soient organiques, métalliques, etc.
ACCESS offrira la possibilité de réaliser des criblages, c’est-à-dire que la plateforme permettra de soumettre des cellules vivantes à des séries de molécules et de dénicher celle qui possède exactement l’effet recherché. Les organismes que les chercheurs utiliseront seront essentiellement la levure Saccharomyces cerevisiae, mais aussi le ver plat Caenorhabditis elegans, la mouche Drosophila melanogaster, les cellules de l’être humain et du hamster.
«L’avantage d’une telle démarche est qu’elle résout deux problèmes d’un coup, explique Howard Riezman. Normalement, ce genre de tests s’effectue dans des éprouvettes, sur des enzymes évoluant hors de leur cellule. Si les chercheurs trouvent une molécule qui remplit leurs critères, leur problème consiste alors à savoir si elle peut entrer dans la cellule et si, une fois à l’intérieur, elle est suffisamment stable pour remplir la tache que l’on attend d’elle. En travaillant directement sur des cellules vivantes, en s’aidant de toute la technologie nécessaire, on s’affranchit de ces deux obstacles.»
Spécialistes des levures
L’essentiel de la plateforme ACCESS sera établi sur le site de l’Ecole polytechnique fédérale de Lausanne qui possède déjà quelques collections de molécules. Une succursale sera toutefois installée à Genève, spécialisée dans l’expérimentation sur des levures. Les chercheurs du Pôle, grâce à l’argent octroyé par la Confédération, s’apprêtent à acheter d’autres collections à travers le monde afin de compléter leur banque qui devrait devenir l’une des plus importantes dans son genre.
Pour augmenter encore cet inventaire gigantesque, un autre projet du PNR vise à exploiter une source plus naturelle et au grand potentiel. L’objectif consiste à chercher, identifier et purifier des molécules synthétisées par des bactéries, des champignons et d’autres plantes qui n’ont pas encore été passés au crible des pharmacologues. Cette contribution est d’autant plus importante que la plupart des agents actifs actuellement sur le marché des médicaments sont justement issus d’organismes naturels.
«La mise en place de la plateforme ACCESS est une priorité et elle devrait s’achever d’ici à un an, précise Howard Riezman. Ensuite, dès qu’elle aura atteint une taille suffisante, nous lancerons des appels d’offres auprès de toutes les équipes scientifiques suisses qui souhaitent réaliser des criblages dans le cadre de leurs recherches, qu’elles soient fondamentales ou dirigées vers une lutte contre une maladie, par exemple. L’idée, à terme, est d’inviter ces équipes à effectuer chez nous cette partie du travail que nous financerons d’ailleurs, grâce à l’argent du Pôle.»
In silico
Et si les molécules d’ACCESS ne suffisent pas, il sera toujours possible de s’en procurer d’autres, virtuelles cette fois. Membre du PRN, Jean-Louis Reymond, professeur au Département de chimie et biochimie de l’Université de Berne est en effet un pionnier dans la synthèse de molécules in silico, c’est-à-dire par ordinateur. En se basant sur les connaissances actuelles en physique et en chimie, ce chercheur s’emploie à construire, atome par atome, des molécules virtuelles dont la forme devrait leur permettre, théoriquement, de se lier à telle protéine, de ressembler à un état de transition de telle enzyme ou encore d’inhiber telle autre substance. Ensuite, le défi des chimistes consistera à la synthétiser pour de vrai et à vérifier si les propriétés définies par ordinateur se concrétisent dans le monde réel.
Les membranes
En plus des processus biologiques, des cascades de réactions et autres kinases, les chercheurs du Pôle focaliseront également leur attention sur les membranes cellulaires. Ces structures essentielles omniprésentes sont en fait assez mal connues. Pendant longtemps, les scientifiques ont pensé qu’elles ressemblaient à une mer de lipide très homogène. En réalité, ils se sont rendu compte ces dernières années que les membranes sont «latéralement inhomogènes». Une fois de plus, les ressources cérébrales du Pôle seront mises à contribution pour concevoir des techniques capables de visualiser ces structures particulières et de déterminer, par exemple, si elles sont souples ou rigides.
«Entre autres choses, les membranes servent bien sûr à éviter que des substances indésirables n’entrent dans les cellules, explique Howard Riezman. Mais parfois, en médecine, on aimerait faire pénétrer à des fins thérapeutiques des molécules qui n’ont normalement pas de droit de passage. Un de nos projets vise justement à trouver le moyen de les faire entrer malgré tout.»
Histoire d’une disciplineC’est l’histoire d’un glissement sémantique qui reflète l’évolution d’une discipline. Au début, elle s’appelait la chimie biologique. En son sein est née la biochimie qui l’a progressivement supplantée. Et aujourd’hui, on voit se développer la biologie chimique. La chimie biologique, en réalité la chimie organique, s’efforçait à l’époque d’isoler, hors des cellules, de grosses molécules comme le glycogène ou l’ADN, dont on voulait déterminer la composition et la structure. Par la suite, petit à petit, l’intérêt scientifique s’est focalisé sur les transformations subies par ces grandes molécules et s’est donc naturellement tourné vers l’activité enzymatique. C’est ainsi qu’a émergé la biochimie. Edmond Fischer est un des pionniers dans ce domaine. Ce biologiste suisse, qui a effectué son collège et son doctorat à Genève avant d’émigrer aux Etats-Unis, a en effet étudié les enzymes responsables de la métabolisation de l’amidon et du glycogène, enzymes qu’il a réussi à reconstituer artificiellement. Et ce sont ses travaux des années 1950 sur la phosphorylation (l’ajout d’un groupe phosphate) réversible des protéines qui lui ont valu le Prix Nobel de physiologie en 1992. La biochimie a alors pris son envol. Son domaine, dès lors, est la cellule en entier. Les biochimistes en dissocient toutes les parties (métabolisme de petites molécules, voies de signalisation diverses…), les étudient séparément avant d’essayer de reconstituer les événements. Les techniques de génie génétique et la découverte du code génétique ont grandement contribué à l’élargissement du champ de recherche de la biochimie. Enfin est venu l’avènement de la biologie chimique. L’idée, cette fois-ci, est d’utiliser les techniques de la biologie, de la chimie et de la génétique pour observer ce qui se passe à l’intérieur d’une cellule maintenue vivante. Les chercheurs genevois du PRN ne sont pas les premiers à réaliser ce genre de recherches. Mais ils comptent bien, à l’image d’Edmond Fischer en son temps, contribuer de manière décisive au développement de cette nouvelle discipline. |
Fiche techniquePôle de recherche national «Biologie chimique» Directeur: Howard Riezman, professeur au Département de biochimie, Faculté des sciences, UNIGE Codirecteur: Kai Johnsson, professeur au Laboratoire d’ingénierie des protéines, Institut des sciences et ingénierie chimiques, EPFL Equipes genevoises participantes: Marcos González-Gaitán, professeur au Département de biochimie et biologie moléculaire, Faculté des sciences Monica Gotta, professeure associée au Département de médecine génétique et développement, Faculté de médecine Jean Gruenberg, professeur au Département de biochimie, Faculté des sciences Robbie Loewith, professeur associé au Département de biologie moléculaire, Faculté des sciences Stefan Matile, professeur au Dépar-tement de chimie organique, Faculté des sciences Aurélien Roux, professeur assistant au Département de biochimie, Faculté des sciences Andreas Zumbuehl, maître-assistant au Département de chimie organique, Faculté des sciences Financement du Fonds national suisse: 13,3 millions de francs sur quatre ans. |
