Un nouveau pipeline d'analyse métagénomique
Le nombre de microorganismes vivant dans notre tube digestif est énorme. Traditionnellement, les cultures bactériennes étaient utilisées pour l'isolement et la classification de nombreuses souches bactériennes. Ces approches se limitent toutefois aux seules bactéries cultivables et permettent une identification à faible résolution de la communauté microbienne.
Le séquençage métagénomique est très prometteur pour les analyses haute-résolution des souches et des sous-espèces, car il évite de devoir cultiver en laboratoire des organismes individuels dans une boîte de Pétri. Cependant, la grande majorité des génomes bactériens ne sont pas encore classifiés et il n'existe aucune information sur le microbiote au niveau des sous-espèces, en partie à cause du manque d'outils bio-informatiques qui permettraient d'analyser l'énorme quantité de données issues du séquençage.
Le pipeline métagénome-ATLAS
Dans un récent article dans BMC Bioinformatics, Silas Kieser du laboratoire du Pr. Mirko Trajkovski laboratory, en collaboration avec le laboratoire du Pr. Evgeny Zdobnov et avec des chercheurs du Pacific Northwest National Laboratory a développé un nouvel outil facile d'utilisation, appelé métagénome-atlas, qui permet d'analyser d'importantes quantités de données. Métagénome-atlas comble une importante lacune en matière d'analyse en combinant les principaux outils actuels. Ce logiciel open-source est facile à installer et à utiliser : les scientifiques peuvent effectuer des analyses métagénomiques avec seulement 3 lignes de commande.
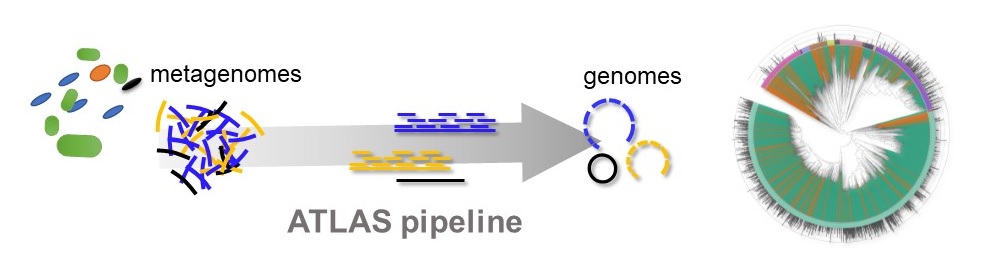
Représentation schématique du pipeline ATLAS qui permet de traiter des séquences génétiques brutes (métagénomes) et de les traduire en nombre de microorganismes (génomes). © unige – Silas Kieser
Un outil éprouvé
Ce nouvel outil est déjà utilisé par les chercheurs du laboratoire du Pr. Mirko Trajkovski pour étudier le microbiome de l'intestin de la souris et ses similarités avec son homologue humain. Il leur a permis d'identifier de nouvelles familles de bactéries, ainsi que des centaines d'espèces et de sous-espèces non-détectables avec la culture traditionnelle en laboratoire. Métagénome-ATLAS est déjà utilisé par des scientifiques du monde entier pour étudier différentes communautés microbiennes allant de l'intestin aux écosystèmes d'eau douce.
