Découvrir de nouveaux médicaments grâce à Darwin
Des chimistes de l’UNIGE ont développé une nouvelle technique de sélection d’assemblages de molécules, permettant de trouver rapidement et à moindres coûts les meilleures combinaisons pour chaque protéine à combattre.
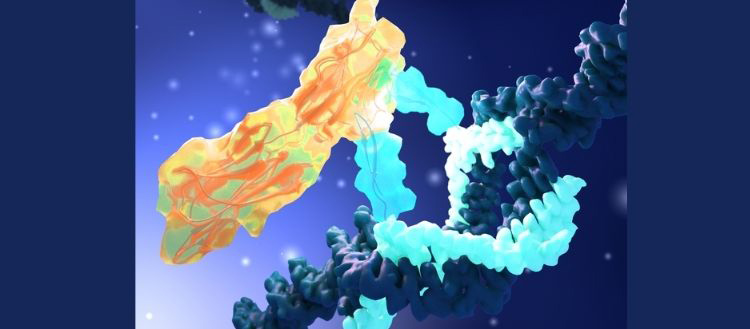
Représentation d’un assemblage de petites molécules (en bleu ciel) reconnaissant la protéine PD-L1 (en jaune).
Notre corps doit sans cesse se défendre contre des bactéries et des virus. Il possède donc des millions d’anticorps différents, amenés à reconnaître l’ennemi et à déclencher la meilleure réponse immunitaire possible. Les scientifiques s’inspirent de ces anticorps pour cibler des protéines à des fins thérapeutiques et perturber leurs fonctions nocives. Toutefois, identifier les petites molécules qui fonderont la base du médicament est un processus long et fastidieux. Des chimistes de l’Université de Genève (UNIGE) ont mis au point une technique inspirée de la théorie de l’évolution darwinienne : l’amplification des meilleures combinaisons et la génération de la diversité permettent à la biologie de trouver une solution à chaque nouveau problème. Ils ont créé une nouvelle méthodologie qui génère rapidement des millions de combinaisons de petites molécules grâce à des processus d’assemblage programmés par l’appariement d’ADN, permettant de trouver en deux semaines la meilleure combinaison possible pour contrer une protéine cible. Ces résultats, à lire dans la revue Nature Chemistry, vont permettre d’ouvrir un nouvel espace encore non exploité pour la création de médicaments.
Le fonctionnement des médicaments est fondé sur la reconnaissance moléculaire d’une protéine cible impliquée dans la maladie, afin de la désamorcer. Pour ce faire, les scientifiques utilisent des criblages à haut débit qui permettent d’identifier quelle molécule pourrait constituer un médicament, en fonction de la protéine visée. Depuis une dizaine d’années, la technique a été améliorée grâce à l’encodage des petites molécules avec des étiquettes ADN qui permettent de simplifier leur identification, l’ADN étant facile à décoder.
S’inspirer des forces évolutives darwiniennes pour trouver des assemblages performants
Aujourd’hui, des chimistes de l’UNIGE sont allé-es plus loin, en s’inspirant des théories de Darwin. «La biologie trouve toujours une solution à un problème, explique Nicolas Winssinger, professeur au Département de chimie organique de la Faculté des sciences de l’UNIGE et auteur de correspondance de l’étude. C’est le principe de l’évolution naturelle qui consiste à amplifier les meilleurs individus, tout en générant de la diversité pour s’adapter et survivre à des conditions changeantes. C’est ce que nous avons mis en place pour les petites molécules.» En effet, les scientifiques ont développé une technologie qui génère de la diversité en créant plus de 100 millions d’assemblages de molécules via leur ADN, qu’ils et elles sélectionnent ensuite pour correspondre au mieux à une protéine particulière.
«Nous nous sommes inspiré-es des caractéristiques des anticorps qui reconnaissent les protéines pour les imiter sous forme de molécules plus simples et permettre ces assemblages en différentes combinaisons, dirigés par les séquences ADN», précise Nicolas Winssinger. Ces combinaisons sont ensuite sélectionnées et amplifiées à plusieurs reprises, afin de trouver la meilleure correspondance possible avec la protéine à combattre, le tout en une à deux semaines, contre des mois voire une année pour des criblages haut-débit traditionnels.
Une technique qui a fait ses preuves, facile à reproduire et peu chère
Afin de valider l’efficacité de cette méthodologie, l’équipe genevoise s’est intéressée à la protéine PD-L1, qui protège les cellules cancéreuses en se faisant passer pour un élément du corps humain et détourner le système immunitaire. «Grâce à notre méthodologie, nous avons identifié rapidement un assemblage qui cible spécifiquement et désamorce PD-L1, confirmant que cela fonctionne avec efficacité», se réjouit Nicolas Winssinger.
Cette technique, simple à reproduire dans n’importe quel laboratoire au monde, n’engage que quelques milliers de francs, contre des millions pour le criblage à haut débit. «En exploitant les forces évolutives mises en exergue par Darwin, nous pouvons dorénavant améliorer nos assemblages de molécules et ouvrir un nouvel espace de combinaisons possibles qui n’a pas encore été exploité, afin de créer de nouveaux médicaments plus performants», conclut le chercheur genevois.
6 déc. 2021
